Palabras clave
INTRODUCCIÓN
La insuficiencia cardiaca es un síndrome complejo, cuya historia natural conduce a la disfunción ventricular izquierda, tanto diastólica como sistólica1, y es una de las principales causas de morbilidad y mortalidad en occidente en general y en España en particular2. Debido al envejecimiento de la población y la mayor longevidad de los enfermos cardiovasculares, la incidencia y la prevalencia de la insuficiencia cardiaca aumentan con la edad3, por lo que, de seguir dicha tendencia, en los próximos 20 años adquirirá proporciones de epidemia, tal como se ha señalado que ocurrirá en nuestro país4. Por otra parte, debido a los elevados costes médicos y sanitarios que conlleva, la insuficiencia cardiaca actualmente es el cuadro cardiovascular más costoso en España5.
A pesar del enorme esfuerzo realizado en los últimos 50 años en la comprensión de la fisiopatología y la farmacología de la insuficiencia cardiaca, diversos estudios recientemente publicados demuestran que todavía existen importantes limitaciones en la prevención y el tratamiento de la insuficiencia cardiaca que explican su mal pronóstico6, por lo que se impone un nuevo enfoque conceptual y práctico de ésta7. En este contexto se inscribe el enfoque «ómico» de la insuficiencia cardiaca.
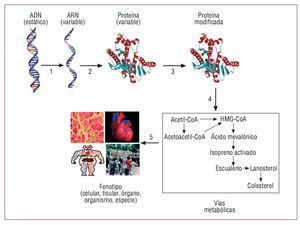
Las «ómicas» son las ciencias biológicas que estudian los genes y sus productos iniciales (transcritos de ARN) y finales (proteínas), así como los productos participantes o derivados (metabolitos) de los procesos metabólicos en los que intervienen las proteínas. La incorporación de la metodología «ómica» al estudio de las enfermedades humanas ha modificado sustancialmente el enfoque biológico de éstas (fig. 1) y ha estimulado enormemente la investigación de nuevos mecanismos, biomarcadores y dianas terapeúticas. Aunque todavía no se conoce con precisión las causas moleculares de la disfunción cardiaca en las enfermedades del corazón que evolucionan a insuficiencia cardiaca, se piensa que son el resultado de alteraciones en la expresión de genes y proteínas. Por ello, se ha propuesto que la combinación de las ciencias «ómicas», principalmente la genómica y la proteómica, puede facilitar la comprensión del origen y el desarrollo de las distintas entidades que configuran el síndrome de la insuficiencia cardiaca, con lo que se propiciaría el establecimiento de perfiles diagnósticos y patrones terapéuticos diferenciales que pueden mejorar el mal pronóstico que la insuficiencia cardiaca conlleva8.
Fig. 1. Escenario de las enfermedades humanas propuesto por las ciencias «ómicas». 1: transcripción; 2: traducción; 3: modificaciones postraduccionales; 4: interacciones; 5: integración.
CONCEPTOS BÁSICOS DE GENÓMICA Y PROTEÓMICA
Genómica
Se denomina genómica al estudio de la organización molecular del ADN y su cartografía física. La genómica engloba distintas subespecialidades. La genómica estructural estudia el plegamiento de las macromoléculas y su estructura tridimensional con la ayuda de técnicas derivadas de la física (p. ej., cristalografía de rayos X) y la bioinformática, y clasifica estas moléculas en familias funcionales. La genómica bioquímica estudia grupos de proteínas específicas y sus correspondientes marcos de lectura abiertos (open reading frames [ORF]). Esto se lleva a cabo mediante su generación y expresión. Por ejemplo, un grupo grande de proteínas de fusión glutatión-ORF se purifican y cartografían para un ORF específico y las proteínas se analizan en mayor profundidad en subgrupos9. La genómica química estudia los efectos de moléculas pequeñas para evaluar sus efectos moduladores en el estado celular o la expresión génica, preferiblemente en sistemas de alto rendimiento10. La genómica funcional o fisiológica se centra en el análisis funcional de la totalidad del genoma (genome wide) y la integración de la estructura del ADN, así como de su función molecular y la interacción de los genes y sus productos génicos11,12. La epigenómica analiza las interacciones entre genomas y proteomas, así como los patrones generales de mutilación y las señales de mutilación, y evalúa este tipo de información en las distintas especies. La genómica comparativa o filogenómica busca determinar el número de familias de proteínas diferentes codificadas por distintos genomas, la distribución de los genes codificantes en los diferentes genomas y cuántos de esos genes los comparten distintos genomas13. La ortogenómica estudia los genomas de descendientes ortólogos, mientras que la paragenómica estudia genomas parálogos. Estos estudios incluyen la composición y la organización de dominios proteínicos en los diferentes organismos. La genómica genética incluye el análisis de perfiles de expresión y de huellas basadas en marcadores en cada individuo en una población que se segrega. La genómica computacional mide cuantitativa o cualitativamente una propiedad de interés, por ejemplo, en varias cepas de ratones. Después se identifican computacionalmente los factores genéticos en regiones genómicas donde el patrón de variación genética se correlaciona con la distribución de rasgos entre las distintas cepas analizadas. El grado de correlación entre los rasgos y los agrupamientos de cepas dentro de cada bloque de haplotipos se evalúa mediante un análisis de la varianza14. La nutrigenómica tiene como objetivo desvelar los efectos de los macronutrientes y los micronutrientes en la salud y la enfermedad en los distintos genotipos15. La toxicogenómica busca entender las complejidades de un sistema biológico en su respuesta a factores tóxicos, mutagénicos o carcinogénicos. Reciben una consideración especial las homologías entre genes encargados de controlar las enfermedades en el genoma humano con respecto a compartir funciones tales como ciclo celular y estructura, adhesión celular, señalización, apoptosis, control neuronal y mecanismos de defensa.
Entre los estudios realizados en el ámbito de la genómica, merece una mención especial la transcriptómica, por su gran trascendencia funcional. La transcriptómica se centra en el estudio del transcriptoma de una célula o un órgano en una situación o una enfermedad determinada. El transcriptoma engloba la colección de ácidos ribonucleicos que se transcriben del genoma, es decir, el perfil de expresión de ARN mensajero (ARNm). En una única célula humana, aproximadamente la mitad de todos los genes puede estar expresándose, y un total de entre 25.000 y 30.000 genes únicos se expresan en el organismo humano. Se ha calculado que el número total de transcritos de estos genes en los diferentes tipos celulares es de 134.135. Mientras que algunos genes se expresan sólo como 0,3 copias por célula, otros poseen hasta 9.417 transcritos.
Tras completar la secuenciación del genoma de los organismos, el interés se centra ahora en la determinación de las funciones de los genes. Este tipo de estudios se puede realizar actualmente gracias al desarrollo de procedimientos de alto rendimiento16. Dichos procedimientos permiten identificar miles de ORF en combinación con los análisis de microarrays. Esta área de estudios integra distintas disciplinas tales como la genética, la biología molecular, la bioquímica, la farmacología (que da lugar a la farmacogenómica o diseño de fármacos que se ajustan mejor a la constitución genética del individuo), la agricultura, la medicina y otras.
Proteómica
La proteómica se define como el estudio del proteoma o conjunto de todas las proteínas presentes en una célula concreta en un momento determinado. El proteoma muestra variaciones dependiendo del estadio de desarrollo, el órgano, el gasto metabólico, la salud del organismo, etc. Puesto que las proteínas se encuentran organizadas y expresadas en sistemas que interactúan, su estudio puede ser muy complicado. Mientras que el genoma no revela los detalles de la función de una célula, el estudio del proteoma tiene exactamente ese objetivo.
En los estudios de proteómica a gran escala se puede detectar la composición/estructura de las proteínas, sus isoformas, los cambios conformacionales, las alteraciones moduladoras durante el desarrollo, las modificaciones postranscripcionales y postraduccionales (fosforilación, glucosilación, etc.), las interacciones con otras proteínas o con fármacos, etc. A partir de cantidades mínimas de compuesto (p. ej., 10 ppm) se puede identificar proteínas individuales en mezclas complejas de miles de moléculas. La proteómica ha modificado la forma de enfocar la investigación de las funciones biológicas. A diferencia del diseño experimental tradicional basado en hipótesis, con la ayuda de las tecnologías ligadas a la proteómica se han hecho posibles aproximaciones más directas, basadas en los patrones de expresión simultánea de redes moleculares de interacción.
Dentro de la proteómica también existen distintas subespecialidades. La proteómica de expresión analiza la proteínas celulares mediante electroforesis bidimensional o cromatografía de alta resolución combinadas con espectrometría de masas17. La proteómica de cartografía celular está interesada en la interacción entre proteínas en distintas fases de la función celular18. La proteómica funcional se centra en el estudio de funciones específicas más que en el proteoma completo19. La proteómica estructural busca comprender la función celular basándose en el análisis tridimensional y la realización de modelos de proteínas20,21. La proteómica inversa comienza con el genoma y procede después con las proteínas.
Relacionada con la proteómica está la metabonómica, que estudia el estado metabólico de fluidos y preparados tisulares de animales empleando espectroscopia de resonancia magnética nuclear de alta resolución en combinación con análisis estadístico. El estudio del conjunto de metabolitos presentes en un momento determinado puede suministrar información relevante sobre el fenotipo de un organismo. Los metabolitos son los verdaderos productos finales de la transcripción génica y reflejan de forma más exacta la actividad celular o fenotipo funcional. Por lo tanto, es probable que el metabonoma sea el «escenario» más adecuado para el estudio de los procesos celulares en situaciones tanto fisiológicas como patológicas22. Los estudios metabonómicos pueden facilitar el desarrollo de tratamientos farmacológicos personalizados si se establecen diferencias del fenotipo metabólico antes y tras la administración de fármacos, puesto que así se puede facilitar la predicción de las respuestas individuales23.
GENÓMICA DE LA INSUFICIENCIA CARDIACA
La insuficiencia cardiaca es ideal para los estudios de genómica, ya que se trata de un síndrome complejo, con múltiples etiologías y factores predisponentes, tanto medioambientales como genéticos. Mientras que la aproximación tradicional, basada en estudiar un pequeño número de genes o un único factor de riesgo, genera una información parcial sobre la insuficiencia cardiaca, la aproximación genómica, que permite identificar un gran número de genes y vías de señalización alteradas en sus distintas fases evolutivas, ofrece una alternativa más eficiente, lo que acelerará el conocimiento de los procesos implicados en su desarrollo24.
Los microarrays de expresión génica suministran información tanto cualitativa (genes activados y genes silenciados) como cuantitativa (nivel de expresión de diferentes genes), lo que permite evaluar cambios sutiles en la expresión génica. El uso de microarrays de expresión génica ha resultado de gran utilidad en el campo de la oncología ayudando al diagnóstico y la clasificación de los tumores25, así como a identificar vías de señalización alteradas, y ha contribuido no sólo a un mejor conocimiento de la fisiopatología de la enfermedad, sino también al desarrollo de terapias más especializadas26. Del mismo modo, la aplicación de los microarrays en la insuficiencia cardiaca puede contribuir a diferenciar subtipos moleculares.
En los estudios en humanos se han utilizado tanto microarrays disponibles comercialmente como diseñados frente a genes cardiacos específicos. Hasta la fecha se han realizado estudios de expresión en pequeñas poblaciones para comparar a pacientes con insuficiencia cardiaca con otros sin ella, y muestras de miocardio antes y después de la implantación de un dispositivo de asistencia ventricular izquierda. Por lo tanto, estos casos representan estadios muy avanzados de insuficiencia cardiaca. La tabla 1 recoge los hallazgos principales de varios de estos trabajos27-44. En dichos trabajos se han encontrado cientos de genes alterados, que están implicados en procesos tales como la señalización mediada por calcio, el metabolismo energético, las vías de apoptosis, la respuesta al estrés, la transducción de señales, el transporte iónico transmembrana y el mantenimiento del citoesqueleto y la matriz extracelular.
Una de las posibles aplicaciones clínicas de este tipo de estudios es su utilidad para la clasificación de las distintas miocardiopatías y la predicción de la respuesta al tratamiento. Así, en pacientes con miocardiopatía dilatada (MCD) se han realizado algunos estudios con el objetivo específico de discriminar la etiología de la insuficiencia cardiaca y definir su perfil de expresión característico34,36,37,44. En este contexto, el análisis de expresión génica ha permitido diferenciar a los pacientes con miocardiopatía ligada al consumo de alcohol y a los pacientes con miocardiopatía familiar32, o entre los pacientes con MCD y los pacientes con miocardiopatía isquémica39. Por otra parte, también se ha demostrado que existen grandes diferencias en el perfil de expresión génica entre pacientes con miocardiopatía hipertrófica y pacientes con MCD en estadios avanzados de la insuficiencia cardiaca43. Asimismo, la utilización de microarrays ha hecho posible discriminar entre el origen isquémico y el no isquémico de la insuficiencia cardiaca con un 89% de sensibilidad y especificidad45. No obstante, en un estudio reciente no se pudo discriminar claramente la cardiopatía de origen isquémico de la no isquémica46. En conjunto, si bien los resultados disponibles parecen prometedores, la utilidad clínica real del empleo de los microarrays de expresión génica para la clasificación y el pronóstico de las miocardiopatías requiere su validación en poblaciones amplias de pacientes, ya que la mayor parte de los estudios publicados hasta la fecha incluyen un número de individuos reducido.
También se está explorando la utilidad de los perfiles de expresión como biomarcadores transcriptómicos pronósticos. En un estudio reciente, en 108 pacientes con MCD de nuevo diagnóstico en el que se comparó a los pacientes con buen y mal pronóstico tras más de 5 años de seguimiento, se ha identificado un panel de 45 genes que están sobreexpresados en los pacientes con buen pronóstico. La determinación de este panel presenta un 74% de sensibilidad y un 90% de especificidad para predecir la evolución de los pacientes47.
Aunque la aproximación farmacogenómica a la insuficiencia cardiaca todavía se halla en sus inicios, la investigación clínica ha demostrado que ciertos polimorfismos neurohumorales modifican la efectividad de los fármacos e influyen en la evolución de los pacientes. Es el caso de los polimorfismos funcionales de genes del sistema renina-angiotensina (SRA), del sistema nervioso simpático (SNS) y de la sintetasa endotelial del óxido nítrico, que afectan a la concentración de efectores de dichos sistemas, así como a sus vías de señalización48.
La evaluación de la información genómica cardiaca no está exenta de limitaciones, en particular que son múltiples los factores que pueden influir en la variabilidad de los resultados obtenidos. Por una parte, la zona del órgano de donde procede el tejido puede influir en los resultados30,49, las variables clínicas como la edad o el sexo también pueden afectar al perfil de expresión génica34,50, y también hay que considerar el impacto de factores como el tratamiento farmacológico, otras enfermedades concomitantes, etc. Por lo tanto, aunque la tecnología de los microarrays y su capacidad para analizar miles de genes simultáneamente están ofreciendo resultados interesantes en algunas áreas, aún hacen falta evidencias más sólidas que respalden su utilización en la práctica clínica general.
PROTEÓMICA DE LA INSUFICIENCIA CARDIACA
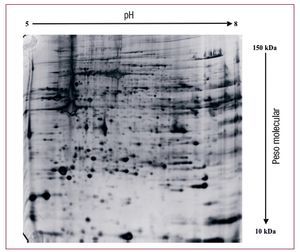
Actualmente existen cuatro bases de datos sobre proteínas cardiacas humanas, basadas en análisis de geles bidimensionales (fig. 2), que han sido establecidas por tres grupos independientes de investigadores y están disponibles en internet. Todas ellas cumplen las normas establecidas para las bases de datos basadas en geles bidimensionales51. Además, se están elaborando bases de datos para otros mamíferos52,53 lo que acelerará el trabajo en modelos animales de insuficiencia cardiaca.
Fig. 2. Imagen de un gel bidimensional de una muestra de ventrículo izquierdo humano teñido con SYPRO Ruby, en la que se observa que las especies de proteínas se localizan a tenor de su peso molecular y su pH.
El análisis de los cambios que se producen en el proteoma del ventrículo izquierdo durante la progresión hacia la insuficiencia cardiaca ha revelado grandes diferencias en el patrón de expresión entre las distintas cardiopatías54-56. No obstante, en su mayoría las proteínas alteradas están relacionadas con los procesos de inflamación, la señalización mediada por calcio, las vías de crecimiento y muerte celular y el citoesqueleto o la matriz extracelular.
La mayoría de los estudios se han centrado en MCD y alguno en miocardiopatía isquémica. Hasta la fecha se han descrito más de 100 proteínas cuya expresión se encuentra alterada en los geles bidimensionales de los pacientes con estas enfermedades; además la mayoría de estas proteínas están disminuidas en los pacientes con insuficiencia cardiaca57-62. Empleando la espectrometría de masas, muchas de esas proteínas se han identificado como proteínas del citoesqueleto y de los miofilamentos, proteínas relacionadas con la mitocondria y la producción de energía y proteínas asociadas con la respuesta al estrés63. Por ejemplo, en el contexto de las proteínas de estrés se han descrito 59 isoformas isoeléctricas de la heat shock protein 27 (HSP27) en el miocardio humano. Doce de estas especies de proteínas se encontraban cuantitativamente alteradas en el corazón de pacientes con MCD e insuficiencia cardiaca, y 10 en el miocardio de pacientes con insuficiencia cardiaca de origen isquémico61.
En lo que se refiere a los estudios en plasma, hay que resaltar los esfuerzos de la plataforma HUPO (Human Proteomic Organisation), cuyo objetivo es analizar de forma sistemática el subproteoma del plasma humano. Los estudios iniciales en insuficiencia cardiaca han detectado alteraciones en las familias de proteínas involucradas en la inflamación, el crecimiento y la diferenciación, la señalización intracelular, el citoesqueleto, los canales/receptores y las proteínas que intervienen en el remodelado del miocardio64. Por otra parte, el análisis del subproteoma del plasma podría tener utilidad pronóstica. Por ejemplo, el análisis de proteínas circulantes en el plasma empleando la tecnología de chips de proteínas SELDI-TOF ha permitido identificar proteínas asociadas al desarrollo de remodelado ventricular izquierdo tras un infarto agudo de miocardio, lo que podría servir para identificar a los pacientes con mayor riesgo de insuficiencia cardiaca asociada al infarto agudo de miocardio65.
Con respecto a la aplicabilidad de la metabonómica en el estudio de la insuficiencia cardiaca, los estudios se centran en la búsqueda de biomarcadores de la evolución de la enfermedad. En un estudio reciente se ha analizado el metaboloma del suero de un grupo de pacientes con insuficiencia cardiaca y fracción de eyección deprimida. De entre los múltiples metabolitos alterados, se seleccionaron el 2-oxoglutarato y la seudouridina, y se observó que la combinación de ambos presentaba más sensibilidad y especificidad que el péptido natriurético cerebral para diagnosticar insuficiencia cardiaca66.
PERSPECTIVAS
Existe la convicción entre los investigadores de que conocer la secuencia completa del genoma y los cambios transcripcionales de miles de genes o conocer la expresión, las modificaciones postraduccionales y las variaciones funcionales de cientos de proteínas no es suficiente para dilucidar la etiopatogenia y la fisiopatología de las enfermedades. Hay un consenso amplio sobre que el verdadero reto radica en el manejo adecuado de tan vasta información.
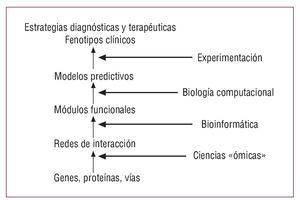
Por ello, tan importante como generar la información «ómica» es desarrollar instrumentos destinados tanto a analizarla eficazmente como a evaluarla con eficiencia. La bioinformática de alto rendimiento y la biología computacional son, respectivamente, los instrumentos que sirven para esos objetivos. Ambos se basan en la biología de sistemas, o sea, la construcción de redes de genes, proteínas y vías metabólicas que interaccionan entre sí para formar módulos funcionales (fig. 3). A su vez, dichos módulos se integran en modelos diseñados para predecir desde los fenotipos clínicos hasta las estrategias diagnósticas y terapéuticas tras la correspondiente experimentación (fig. 3).
Fig. 3. Etapas del proceso de la biología de sistemas, desde la generación de datos moleculares y bioquímicos hasta la evaluación y la validación experimental básica y clínica.
La biología de sistemas del corazón se halla todavía en su infancia67. Hasta el presente se han desarrollado modelos que integran datos parciales del genoma, el proteoma y el funcionamiento de organelas subcelulares como la mitocondria68 o del cardiomiocito entero69. En lo que concierne a la insuficiencia cardiaca, ya se ha efectuado el primer intento de sistematización elaborando modelos diagnósticos clínicos a partir de los cambios en la expresión génica detectados en muestras de pacientes con MCD u otras cardiopatías70. Cabe pensar que en un futuro cercano se dispondrá de más modelos holísticos que integrarán desde el genoma cardiaco hasta el comportamiento de las cardiopatías y harán posible el «manejo personalizado» de los pacientes con insuficiencia cardiaca.
Full English text available from: www.revespcardiol.org
Sección patrocinada por el Laboratorio Dr. Esteve
Correspondencia:
Dr. J. Díez.
Área de Ciencias Cardiovasculares. Edificio CIMA. Avda. Pío XII, 55. 31008 Pamplona. Navarra. España.
Correo electrónico: jadimar@unav.es