Con más de un 20% de las muertes de lactantes y alrededor de un 40% de las muertes prenatales cada año, las cardiopatías congénitas (CC) son los trastornos congénitos más comunes en el recién nacido y, por lo tanto, suponen una carga creciente para los sistemas de asistencia sanitaria1. Además, está en aumento el número de niños afectados por CC, incluso de las formas más complejas, que sobreviven hasta la edad adulta, con lo que superan la cohorte de las CC pediátricas y requieren asistencia cardiaca especializada durante toda la vida1. En consecuencia, hay necesidad urgente de profundizar en el conocimiento de los mecanismos genéticos y moleculares relacionados con la enfermedad, con objeto de establecer nuevas estrategias preventivas y tratamientos.
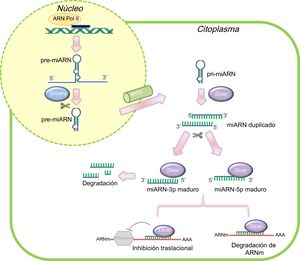
Varias décadas de investigación intensiva han revelado las complejas redes reguladoras que gobiernan el desarrollo cardiovascular y han subrayado la importancia de los factores genéticos, así como de los estímulos ambientales, en la patogenia de la CC. Recientemente ha surgido un nuevo mecanismo en el que interviene una regulación postranscripcional a través de micro-ARN (miARN) como elemento central del desarrollo cardiaco embrionario2. Los miARN son una clase de ARN reguladores pequeños y conservados en el proceso evolutivo, de aproximadamente 20 a 26 nucleótidos de longitud en la fase madura. Estas moléculas son de acción postranscripcional, fundamentalmente mediante un emparejamiento de bases con el ARNm diana imperfecto y específico de la secuencia, con lo que se produce represión traslacional y silenciamiento génico (figura 1)3.
Representación esquemática de la biogénesis de los micro-ARN (miARN) y su modo de acción en las células de mamíferos. En la vía canónica de la biogénesis de los miARN, los pri-miARN se transcriben en el núcleo por la acción de la ARN polimerasa II y son procesados por el Drosha para dar lugar a un precursor más corto de ∼65 nucleótidos de longitud (pre-miARN), el cual es exportado luego al exterior del núcleo. En el citoplasma, el Dicer reconoce y fragmenta las moléculas de pre-miARN, produciendo con ello estructuras dúplex de miARN de ∼22 nucleótidos. Habitualmente, el brazo con el extremo 5’ más libre se incorpora de manera preferente al RISC con objeto de silenciar la diana de ARNm. No obstante, continúa la controversia respecto a la función biológica de la otra hebra. El emparejamiento complementario entre ARNm y miARN maduro determina el mecanismo de silenciación génica. ARNm: ARN mensajero.
Según indican los estudios publicados hasta la fecha, diversas formas de CC se asocian con alteraciones en la expresión de miARN específicos y sus genes objetivo, lo cual indica que una regulación adecuada de la expresión del miARN durante la cardiogénesis podría ser crucial para la prevención de las CC. Durante las últimas décadas, el desciframiento de la significación de la regulación postranscripcional mediada por miARN en la patología de las CC se ha beneficiado de amplios estudios en diferentes modelos que han aportado nuevas perspectivas respecto al desarrollo cardiaco. Concretamente se ha examinado la deleción, en tejidos específicos, de enzimas que son esenciales para la biogénesis de los miARN (p. ej., Dicer, Drosha, Ago2, DGCR8) en modelos murinos y de pez cebra. De hecho, en los modelos knock-out se produjo una mortandad embrionaria temprana debida a una detención general del crecimiento y defectos del desarrollo cardiaco, lo cual pone de manifiesto la importancia de los miARN en el sistema cardiovascular3. Este papel se ha confirmado también en experimentos de ganancia o pérdida de función, que indican que un equilibrio fino en la cantidad de miARN presente es fundamental para que se mantenga la homeostasis cardiaca y que una desviación respecto a ese equilibrio puede ser de gran importancia en la enfermedad cardiovascular4.
El músculo cardiaco es rico en numerosos miARN, como miR-1, miR-133, miR-499 y miR-208, que se expresan de manera abundante, aunque no exclusiva, en el tejido cardiaco. Los estudios realizados en el corazón en desarrollo de ratones y de Xenopus laevis han mostrado que una expresión errónea de estos miARN específicos del músculo altera la formación del tejido cardiaco, lo que da lugar a una pared ventricular delgada y muerte embrionaria temprana a causa de malformaciones cardiacas graves5. Las recientes tecnologías de alto rendimiento, que combinan análisis bioinformáticos y moleculares, han establecido claramente la potencia de estos miARN para modular, e incluso controlar, factores de transcripción cardiacos fundamentales como Gata4, MEF2C, Tbx1, genes de la cadena pesada de miosina cardiaca y SRF, cuya desregulación se ha asociado anteriormente con el desarrollo de las CC4.
Se han analizado funcionalmente otros miARN de expresión ubicua, como miR-17-92, miR-195, miR-196a y miR-363, y se ha demostrado que participan en el control de la diferenciación del miocardio durante la embriogénesis de los mamíferos. De hecho, otras pruebas obtenidas in vivo han demostrado que la alteración de la expresión de estos miARN puede inducir malformaciones cardiacas conotruncales y/o comunicaciones interventriculares en distintos modelos animales, probablemente a través de la represión de genes progenitores cardiacos clave, como Isl1, Tbx1, Hoxb8 y Hand16.
A este respecto, resulta sorprendente que los miARN puedan actuar a través de bucles de retroalimentación, con lo cual regulan su propia expresión y la de sus miARN diana. Este es el caso de miR-1 en las células musculares, que puede modificar la expresión de la histona desacetilasa 4 (HDAC4), un represor del factor de transcripción MEF2 que, a su vez, activa el miR-1 y otros miARN diana4,7. Este bucle de retroalimentación de regulación fina podría tener un papel clave en la proliferación y la diferenciación celulares y con ello probablemente contribuya a causar trastornos estructurales o funcionales relacionados con el corazón.
Considerados en conjunto, los resultados de los estudios realizados in vivo e in vitro han sentado las bases para la realización de estudios en el ser humano, al caracterizar una amplia variedad de miARN que podrían participar en el desarrollo de las CC en el ser humano cuando se altera su regulación.
Es de destacar que la desregulación de los patrones de expresión de los miARN en el ser humano se ha documentado predominantemente en enfermedades cianóticas y comunicaciones interventriculares, y se ha resaltado la presencia de un perfil alterado de miARN, principalmente en el tejido miocárdico humano. Concretamente, se ha descrito que múltiples miARN con una expresión diferenciada, como miR-1, miR-206, miR-421, miR-940, miR-181c y miR-138, se asocian con tetralogía de Fallot y defectos del tabique, que son las formas más frecuentes de CC8,9. Además, recientemente se ha identificado un perfil de expresión de miARN atípico en el miocardio del ventrículo derecho de pacientes con síndrome de corazón izquierdo hipoplásico; ello afecta a la expresión de varios genes diana que son importantes para el adecuado desarrollo cardiaco10.
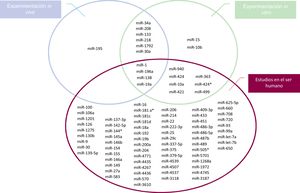
En la figura 2 se resume la expresión de miARN aberrante específica observada en las investigaciones experimentales y en humanos. Aunque los resultados de todos estos estudios han aportado un fundamento para investigar más detalladamente el potencial de los miARN durante el desarrollo cardiaco, la determinación del perfil de estos miARN en la CC humana conlleva algunas dificultades intrínsecas. De hecho, la amplia heterogeneidad de la expresión de miARN que se deduce de todos estos estudios refleja la etiología compleja de las CC y la elevada heterogeneidad genética entre los pacientes con CC. Por otra parte, los miARN identificados pueden estar sesgados debido al subtipo de CC, el pequeño tamaño muestral y los diferentes materiales utilizados para el análisis (p. ej., líneas celulares, tejido cardiaco o sangre). Por fortuna, muchas de estas limitaciones se han superado gracias a la reciente introducción de las técnicas de alto rendimiento, y hoy se dispone de una amplia gama de métodos centrados en la sensibilidad y la especificidad para la detección de miARN.
El creciente interés por el perfil de miARN y su asociación con diferentes tipos de CC ha sentado las bases para la búsqueda del papel de los miARN circulantes como posibles biomarcadores diagnósticos y pronósticos. De hecho, dada su mayor estabilidad en la sangre y el suero, se podría usar los miARN liberados a la circulación como biomarcadores no invasivos para la detección sistemática prenatal y la evaluación del pronóstico. Hasta la fecha, tan solo unos pocos estudios piloto han explorado la diferencia de expresión de los miARN en la sangre y el suero de los pacientes con CC, por lo que es prioritario explorar con mayor detalle su posible aplicación en el ámbito clínico9. Tiene interés señalar que algunos datos han correlacionado con la CC la expresión de los miARN placentarios en el suero de las gestantes, lo cual apunta a nuevas posibilidades por lo que respecta a las pruebas prenatales no invasivas para la detección de las CC11.
Recientemente se ha propuesto otra hipótesis respecto al efecto de las variantes genéticas en los genes de miARN (es decir, pri-miARN, pre-miARN y miARN maduros) en la biogénesis y la función de miARN específicos12. De hecho, los polimorfismos de nucleótido único (SNP) de miARN de ganancia y pérdida de función pueden afectar al procesamiento y la posterior maduración de los miARN, así como a la selección de dianas de miARN, lo cual influye en el riesgo o el pronóstico de diversas enfermedades humanas, incluidos varios trastornos cardiacos.
Por lo que respecta a la susceptibilidad a las CC, el SNP rs11614913 en el miR-196a2, situado en la secuencia pre-miARN, parece ser el más prometedor, si bien sus consecuencias biológicas en el desarrollo del corazón continúan siendo controvertidas y se requerirán nuevos estudios para dilucidarlas13. No obstante, hasta ahora son pocas las variantes de los genes de miARN que se han asociado con riesgo de CC, y la relación entre los SNP de miARN y las CC continúa siendo en gran parte desconocida. Estas observaciones son coherentes con el hecho de que las variantes genéticas en los genes de miARN se produzcan con poca frecuencia (alrededor del 10% en la secuencia de pre-miARN y menos del 1% en la región de siembra), y ello resalta la rigurosa preservación de la identidad de secuencia de siembra en una misma especie y entre diferentes especies.
En línea con esta observación, la presencia de SNP en la región no traducida (UTR) 3’ de los genes diana de miARN ha generado mucho interés, y se ha observado también que desempeña un papel en algunas formas de CC. Así ocurre con los SNP rs3203358 y rs6489956, situados en las 3’UTR de GATA4 y Tbx5 respectivamente. Estos genes codifican 2 de los factores de transcripción mejor caracterizados, que son cruciales para el desarrollo normal del corazón. De hecho, 2 elegantes estudios han demostrado un efecto significativo de estas 2 variantes en el aumento de la susceptibilidad a las CC al afectar a la capacidad de unión de miARN específicos14,15. Hasta la fecha, aunque su relevancia es indudable, tan solo se han realizado unos pocos estudios en los que se haya investigado la posible repercusión de los SNP relacionados con miARN en el riesgo de CC. Los enfoques comprehensivos e integradores pueden ser útiles para comprender mejor los efectos de las variantes genéticas en el circuito regulador de las células dirigido por miARN específicos, cuya desregulación podría conducir a malformaciones cardiacas estructurales y funcionales. De hecho, la investigación de qué genes y vías se ven afectados puede conducir a un conocimiento más profundo de los mecanismos moleculares que intervienen en la cardiogénesis en condiciones tanto fisiológicas como patológicas, y es posible que ello identifique en última instancia dianas contra las que se pueda actuar para desarrollar tratamientos basados en miARN específicos.
Aunque el estudio de los miARN en la patogenia de las CC es un ámbito aún relativamente nuevo, estas moléculas son muy prometedoras como factores cruciales en el desarrollo del corazón que bridan nuevas oportunidades para esclarecer mejor la amplia heterogeneidad de la CC y mejorar su diagnóstico prenatal.
CONFLICTO DE INTERESESNo se declara ninguno.